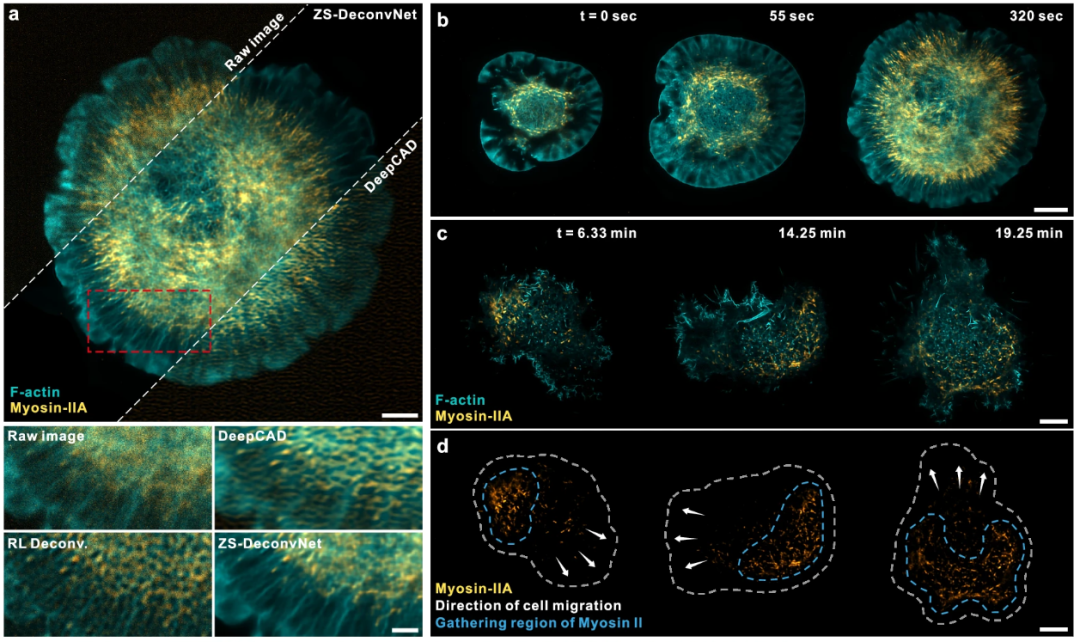
图示:通过 ZS-DeconvNet 对快速光敏生物过程进行长期 SR 成像。(来源:论文)
编辑 | 萝卜皮
计算超分辨率方法,包括传统的分析算法和深度学习模型,极大地改进了光学显微镜。其中,有监督深度神经网络表现出了出色的性能,但由于活细胞的高动态性,需要大量的高质量训练数据,而获取这些数据非常费力甚至不切实际。
在最新的研究中,清华大学和中国科学院的研究人员开发了零样本反卷积网络(Zero-shot deconvolution networks,ZS-DeconvNet),可立即将显微镜图像的分辨率提高超过衍射极限 1.5 倍以上,同时荧光比普通超分辨率成像条件低 10 倍,以无监督的方式进行,无需地面事实或额外的数据采集。
研究人员还展示了 ZS-DeconvNet 在多种成像模式上的多功能适用性,包括全内反射荧光显微镜、三维宽视场显微镜、共焦显微镜、双光子显微镜、晶格光片显微镜和多模态结构照明显微镜;它能够对从有丝分裂单细胞到小鼠和秀丽隐杆线虫的多细胞胚胎的亚细胞生物过程进行多色、长期、超分辨率 2D/3D 成像。
该研究以「Zero-shot learning enables instant denoising and super-resolution in optical fluorescence microscopy」为题,于 2024 年 5 月 16 日发布在《Nature Communications》。

光学荧光显微镜对于生物研究至关重要,而超分辨率技术的进步极大提高了成像细节,但伴随空间分辨率提升的是其他成像参数的折衷。计算超分辨率方法凭借其能在线提升图像质量、增强现有设备性能且扩展应用范围的能力,成为研究热点。
这些方法分为基于分析模型的去卷积等技术和基于深度学习的 Super-Resolution(SR)网络两大类。前者受限于参数调优及对复杂成像环境适应性差的问题,后者虽能通过大数据学习复杂图像转换,却面临数据获取难度大、高度依赖训练数据质量的挑战,这限制了深度学习超分辨率技术在生物研究日常应用中的普及。
在这里,清华大学和中国科学院的研究团队提出了一个零样本反卷积深度神经网络框架 ZS-DeconvNet,它能够以无监督的方式训练 DLSR 网络,仅使用一张低分辨率和低信噪比的平面图像或体积图像堆栈,从而实现零样本实现。
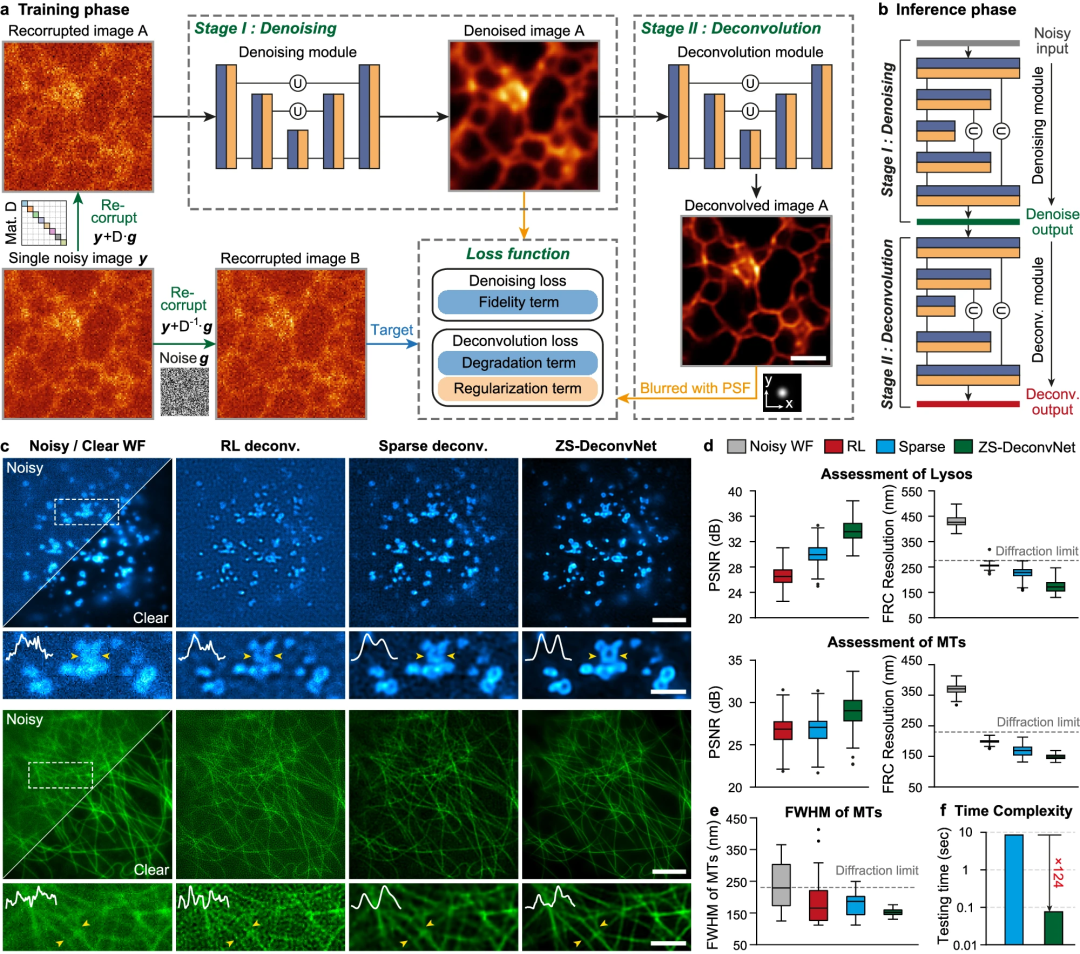
图示:零样本反卷积网络。(来源:论文)
因此,与最先进的 DLSR 方法相比,ZS-DeconvNet 可以适应不同的生物成像环境,其中生物过程过于动态、对光过于敏感而无法获取真实的 SR 图像,或者图像获取过程受到未知和非理想因素的影响。
研究人员表示,即使在单个低信噪比输入图像上进行训练,ZS-DeconvNet 也可以将分辨率提高超过衍射极限 1.5 倍以上,并具有高保真度和可量化性,并且无需进行特定于图像的参数调整。
ZS-DeconvNet 适用于多种成像模式,从扫描显微镜到宽场检测显微镜,并在多种样本和显微镜设置中展示了其能力。
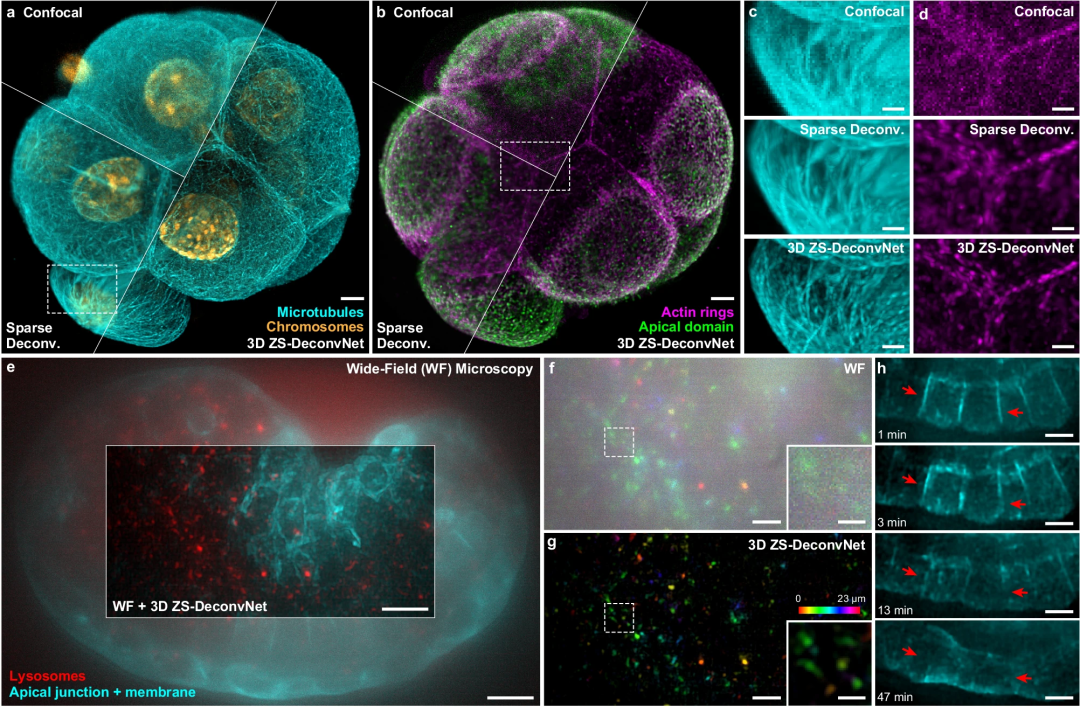
图示:将 ZS-DeconvNet 推广到多种成像模式。(来源:论文)
研究人员证明了经过适当训练的 ZS-DeconvNet 可以在毫秒时间尺度上推断出高分辨率图像,实现对多个细胞器相互作用、迁移和有丝分裂的光敏感过程中的细胞骨架和细胞器动力学,以及发育中的线虫和小鼠胚胎的亚细胞结构和动力学的高通量长期 SR 2D/3D 成像。
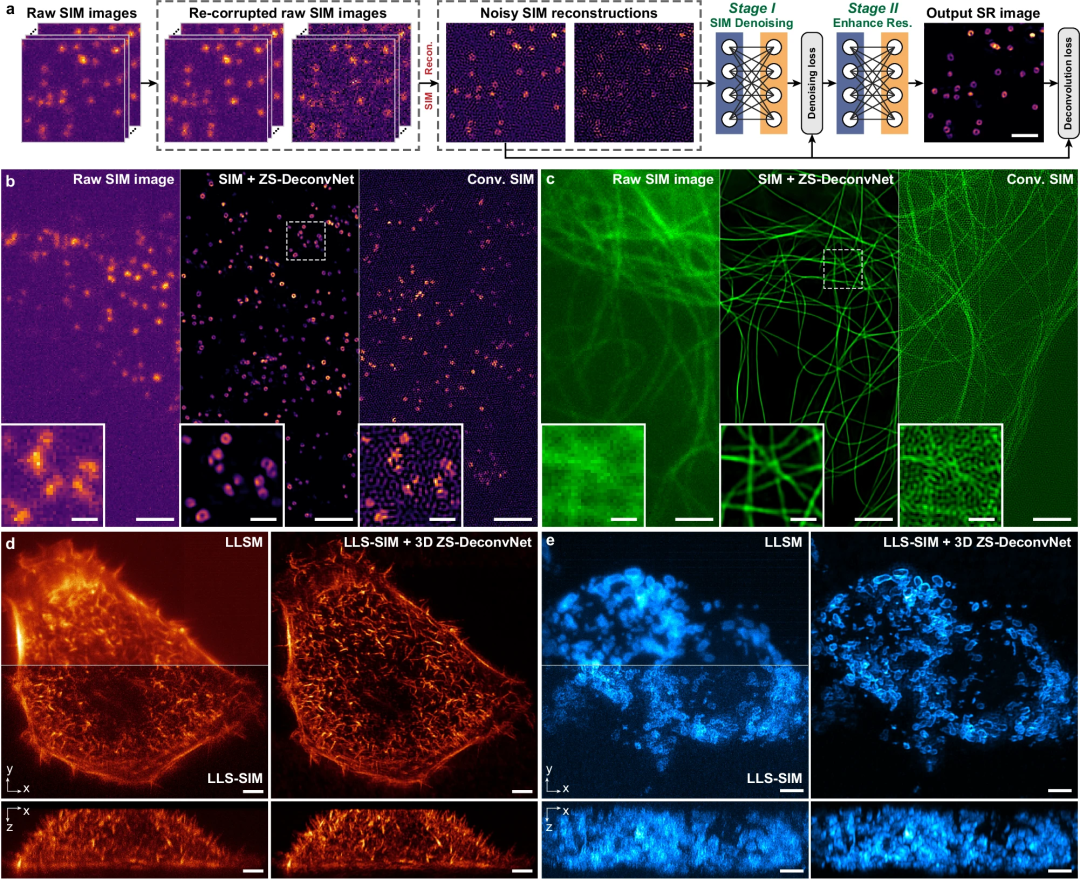
图示:多模态 SIM 数据中的零样本去噪和分辨率增强。(来源:论文)
此外,为了让生物学研究社区能够广泛使用 ZS-DeconvNet,该团队建立了一个 Fiji 插件工具箱和一个 ZS-DeconvNet 方法的教程主页,用户无须深度学习知识也能轻松使用。
尽管具有广泛适用性和稳健性,ZS-DeconvNet 用户需注意潜在的幻想生成及其局限,如低荧光信号误识别、应用于不同成像模式的图像时性能下降、PSF 匹配不当导致的问题,以及无监督学习下的分辨率提升不如监督学习明显。
未来,通过结合更先进的网络架构、拓展至其他光学超分辨技术、采用领域适应或泛化技术,以及处理空间变化的PSF,ZS-DeconvNet的功能和应用范围将进一步扩大。
论文链接:https://www.nature.com/articles/s41467-024-48575-9



