编辑 | 白菜叶
生物网络通过详细描绘基因、蛋白质及其他细胞成分之间的复杂相互作用,为建模生物系统提供了重要工具。
这些网络将实体表示为节点,将其相互作用(从物理连接到功能关联)表示为边,从而为解析生物系统和过程的复杂性奠定了基础。
例如,在蛋白质-蛋白质相互作用(PPI)网络中,复杂的连接关系包含了理解细胞过程和疾病机制的关键信息。然而,解读这些网络以获取生物学洞察仍面临巨大挑战。
在最新的研究中,斯坦福大学(Stanford University)的研究人员提出了一种自监督网络嵌入框架,称为判别网络嵌入 (DNE,discriminative network embedding)。
与主要关注直接或有限阶节点邻近度的传统方法不同,DNE 通过利用邻近节点和远距离节点的表示之间的对比来在本地和全局上表征节点。
测试显示 DNE 在各种关键网络分析(包括 PPI 推理和蛋白质功能模块的识别)中均比现有技术表现优异。
该研究以「Deep representation learning of protein-protein interaction networks for enhanced pattern discovery」为题,于 2024 年 12 月 18 日发布在《Science Advance》。

蛋白质-蛋白质相互作用 (PPI) 网络是理解生物系统动态的基础,其中节点代表蛋白质,边代表蛋白质之间的无数相互作用。
虽然它们在现代生物学中发挥着关键作用,但从这些相互交织的网络中可靠地辨别模式仍然是一项艰巨的挑战。
挑战的本质在于全面描述网络中每个节点与其他节点的关系,并有效地利用这些信息进行准确的模式发现。
新方法 DNE
最新的研究中,斯坦福大学的研究人员介绍了一个通用的图表示学习框架,该框架使用深度学习在低维空间中保留网络的非线性和多面结构,以便对生物网络进行高性能分析。该方法称为判别网络嵌入 (DNE),通过其直接相邻和网络中较远节点的表示之间的非线性对比来表征每个节点。
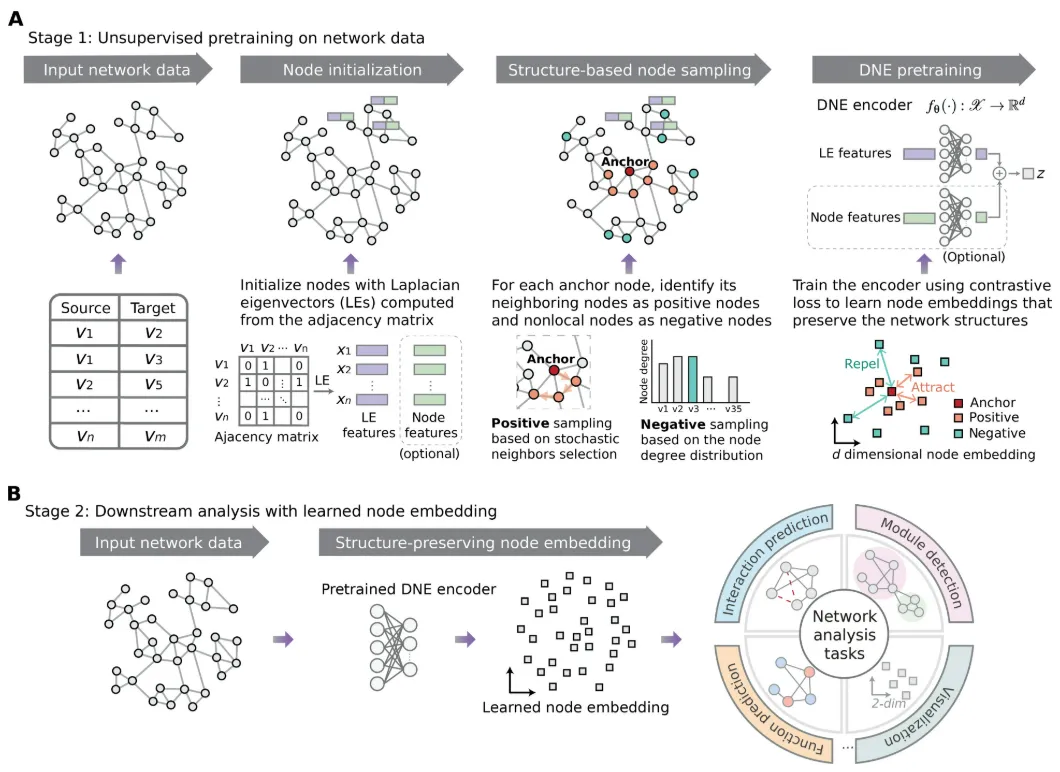
图示:DNE 概述。(来源:论文)
DNE 方法允许从整体上看待网络中每个节点的作用:它突出显示节点的直接连接,例如 PPI 网络中蛋白质之间的相互作用,以及网络内的社区关系,例如蛋白质功能模块。
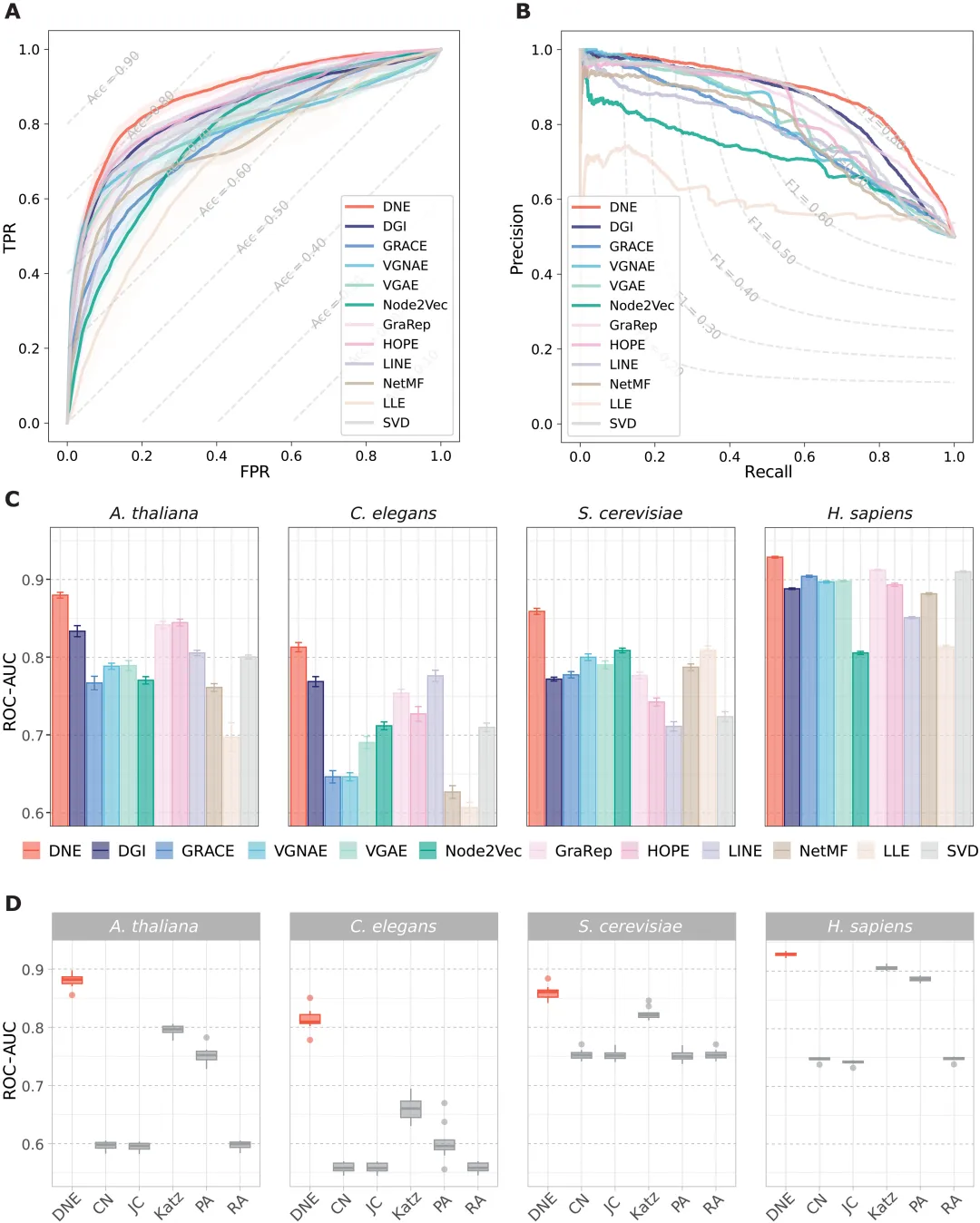
图示:四个 PPI 基准中不同链接预测方法的表现。(来源:论文)
与主要关注节点间有限阶接近度的传统方法不同,DNE 根据其近邻和较远节点的表示之间的对比来表征每个节点。通过同时考虑局部连接模式和与更广泛网络的交互,DNE 可以更全面地了解网络内的节点关系。
研究人员在多个 PPI 数据集上对 DNE 的评估表明,它在准确预测 PPI 和识别功能模块方面的能力优于现有方法。DNE 还表现出对网络扰动的稳健性,并且在不同的扰动率下始终优于其他方法。
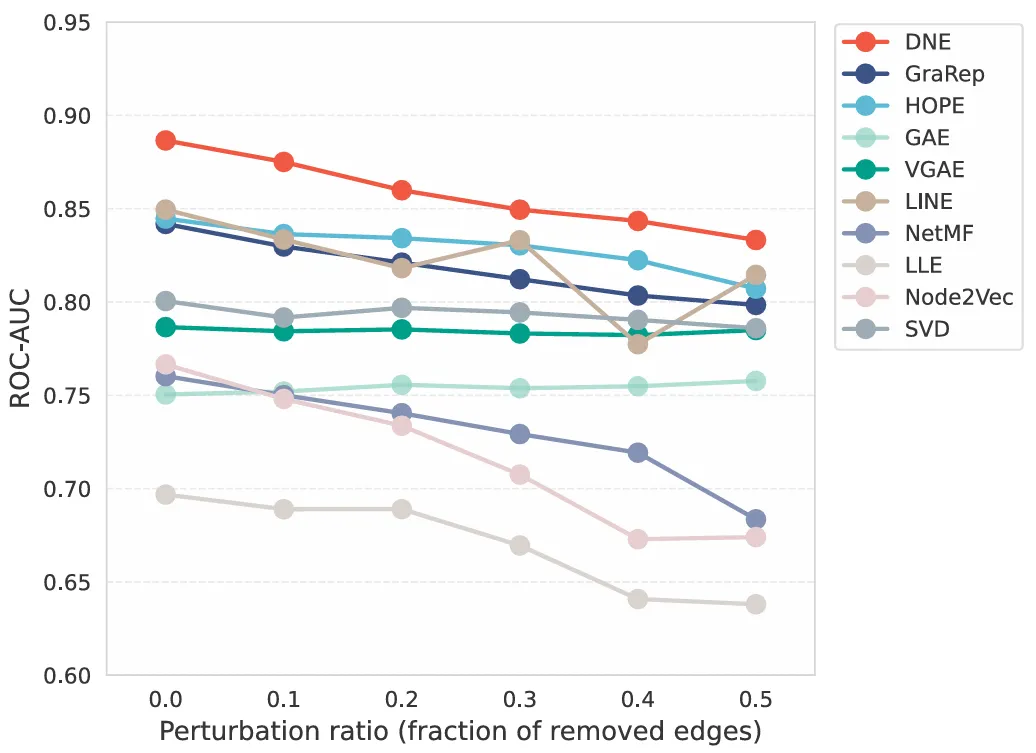
图示:稳健性评估显示,DNE 的 ROC-AUC 分数与其他九种网络嵌入方法相比,可抵抗链接扰动,其中链接以不同的比率随机删除。(来源:论文)
同时,DNE 通过其嵌入反映蛋白质在 PPI n 跳距离和基因本体功能相似性方面的接近度,有效地捕获了具有生物学意义的信号。
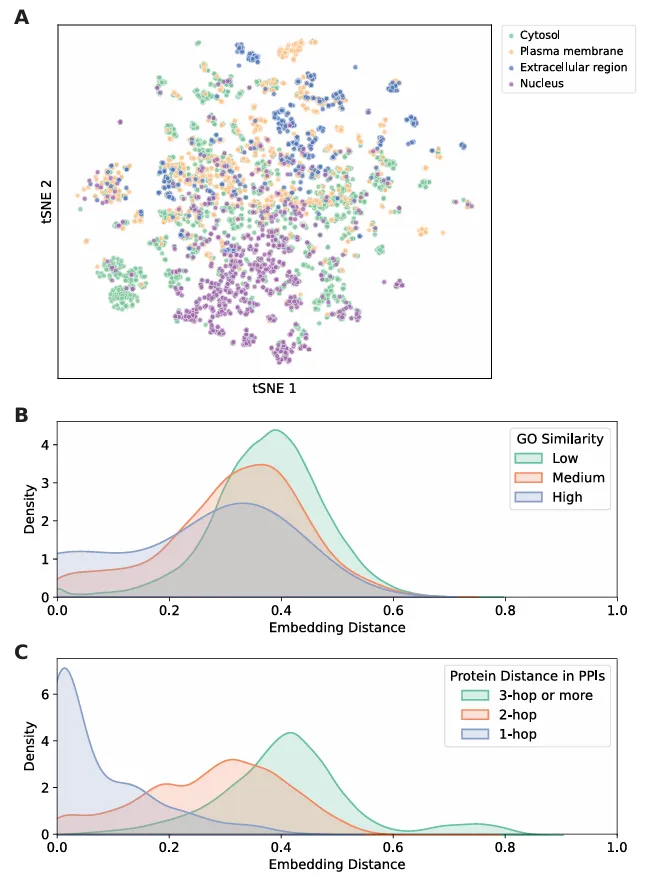
图示:DNE 蛋白质嵌入的可视化。(来源:论文)
虽然 DNE 能够仅从网络的结构信息中得出节点嵌入,但当这些特征可用时,它还可以灵活地将节点特征合并到嵌入过程中。
在像 PPI 这样的生物网络中,每个节点代表一种蛋白质,节点(或蛋白质)特征可以来自多种来源,例如氨基酸序列、蛋白质的三维(3D)结构和蛋白质定位,从而提供网络内蛋白质拓扑功能之外的附加信息。
DNE 方法通过整合来自预训练蛋白质语言模型的蛋白质序列特征来增强网络嵌入,为预测 PPI 提供了一种卓越的方法。与仅依赖序列数据的现有方法相比,这种整合大大提高了 PPI 预测准确性。
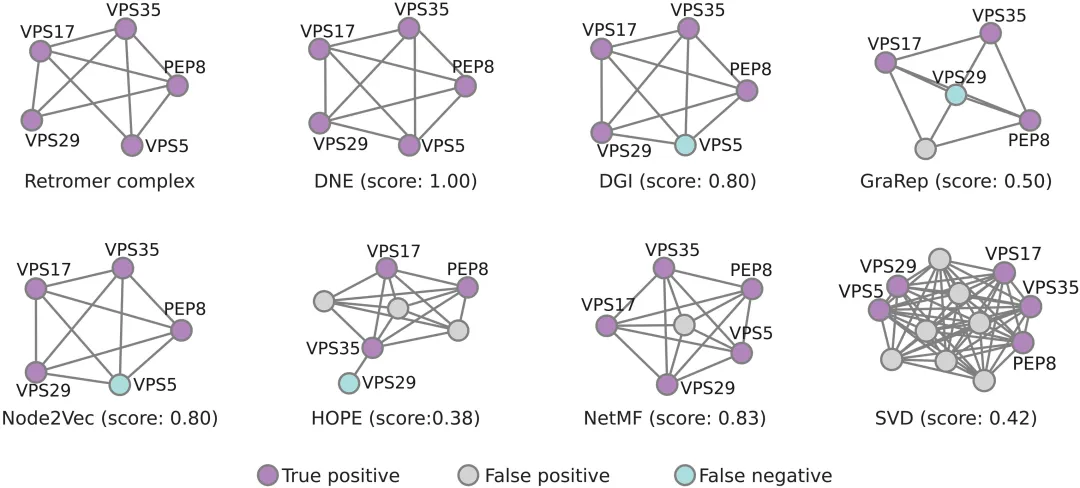
图示:评估预测复合物与标准 Retromer 复合物之间的重叠。(来源:论文)
DNE 的优势
总体而言,DNE 为网络分析提供了几个优势。
第一,它生成更具判别性的嵌入,不仅可以捕获每个节点的局部连接模式,还可以将这些模式与网络其他部分的模式区分开来。这可以更准确地表示每个节点的结构角色和社区成员身份,从而降低过度拟合局部网络噪声的可能性。
第二,通过整合来自近邻以及其他网络段的数据,DNE 提供了整个网络的更全面的视图。
第三,DNE 可以利用网络结构和节点特征来生成更丰富的嵌入。
研究中,这些嵌入用于推断蛋白质相互作用并识别功能模块。进一步的应用可能包括疾病基因预测,其中嵌入有助于识别与疾病机制相关的蛋白质,以及蛋白质功能预测,以促进对新测序基因组中的蛋白质进行注释。
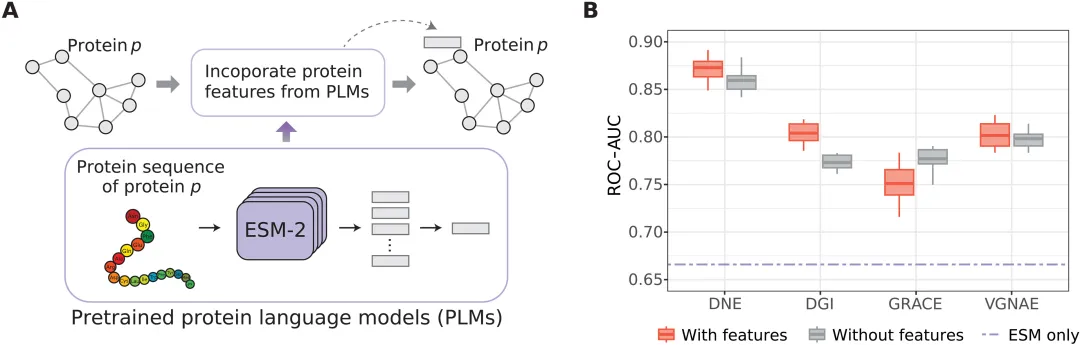
图示:结合蛋白质特征的链接预测中各种方法的性能比较。(来源:论文)
值得注意的是,DNE 的适用范围不仅限于 PPI 网络,还适用于各种领域。对引文网络、电网和互联网服务提供商网络等各种网络类型的初步研究结果表明,DNE 具有更广泛的适用性。
研究人员在论文里表示,他们所提出的方法标志着网络嵌入的显著进步,并为高性能网络分析提供了迫切需要的解决方案。
改进空间
虽然所提出的方法在网络分析方面前景光明,但未来仍有改进空间。
首先,该方法目前优先考虑结构信息而不是节点特征。虽然 DNE 可以整合节点特征,但它们主要用于初始化嵌入,以便最终嵌入可以反映这些节点属性。通过在上下文节点采样期间考虑节点特征之间的相似性以及节点连接,可以改进此过程。
其次,所提出的方法使用多层感知器 (MLP) 作为编码器。研究其他网络类型以用作编码器也可能很有趣,例如图神经网络。诸如 PPI 之类的生物网络是推进科学家对复杂生物系统理解的支柱。然而,它们固有的复杂性往往会给分析带来挑战,并阻碍下游应用。
结语
总而言之,该团队提出了一种自监督网络嵌入技术,旨在为高维网络数据提供更具辨别力的低维嵌入。所提出的技术通过利用每个节点的本地环境和更广泛的网络环境的见解,以独特的方式捕捉每个节点的内在特征。
针对各种生物网络的大量实验研究表明,这种双重视角提供了全面而稳健的网络表示,从而实现了可靠的模式发现和准确的下游网络分析。
因此,DNE 有望成为生物信息学和系统生物学领域的宝贵工具。
论文链接:https://www.science.org/doi/10.1126/sciadv.adq4324



